ChIP+SIM看减数分裂过程中的RPA、RAD51、DMC1
来源:智能网
时间:2020-09-15 18:08:53
热度:253
ChIP+SIM看减数分裂过程中的RPA、RAD51、DMC1之前我们整理分享过一系列关于减数分裂的文章,最近又上线一篇,将ChIP-seq、SIM超分辨两大技术联合使用,追踪减数
之前我们整理分享过一系列关于减数分裂的文章,最近又上线一篇,将ChIP-seq、SIM超分辨两大技术联合使用,追踪减数分裂中的关键事件DNA损伤修复,具体内容如下:
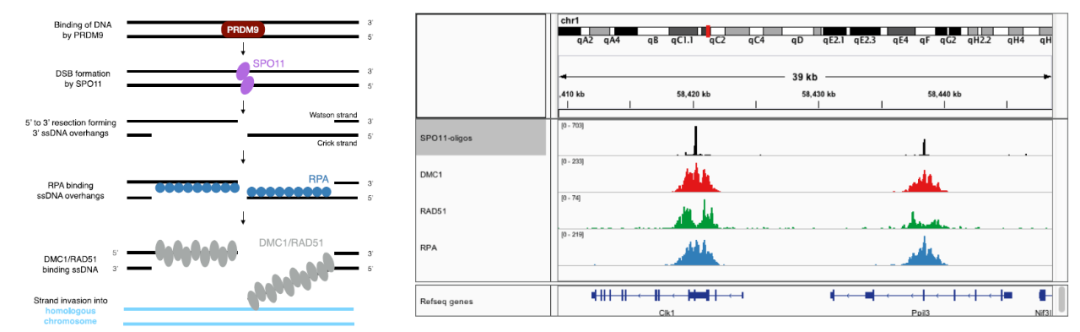
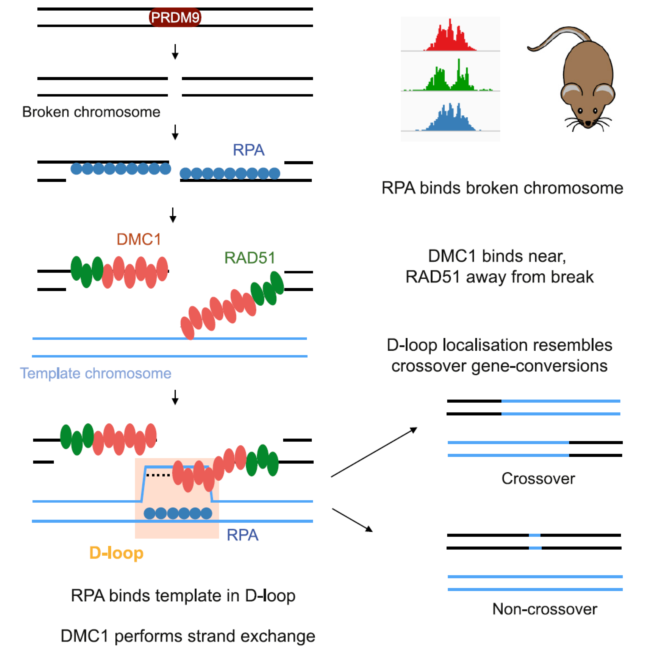
先搭建模型:减数分裂过程中,组蛋白甲基转移酶PRDM9修饰组蛋白,“标记”损伤发生的位点,SPO11切割DNA,引发DNA双链损伤(DSB)。DSB发生后RPA、DMC1、RAD51参与DSB修复,作者构建了一种杂交小鼠,可识别区分出小鼠两条同源染色体(便于追踪同源介导修复过程中的损伤染色体、模板染色体),利用ChIP-seq技术追踪DSB修复过程中RPA、DMC1、RAD51在DNA序列上的分布情况
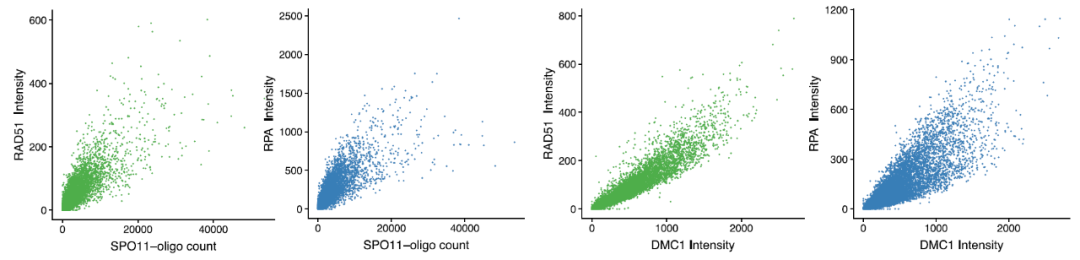
宽泛的看一下DSB数(SPO11指示)、RPA、DMC1、RAD51之间的关系,发现它们正相关
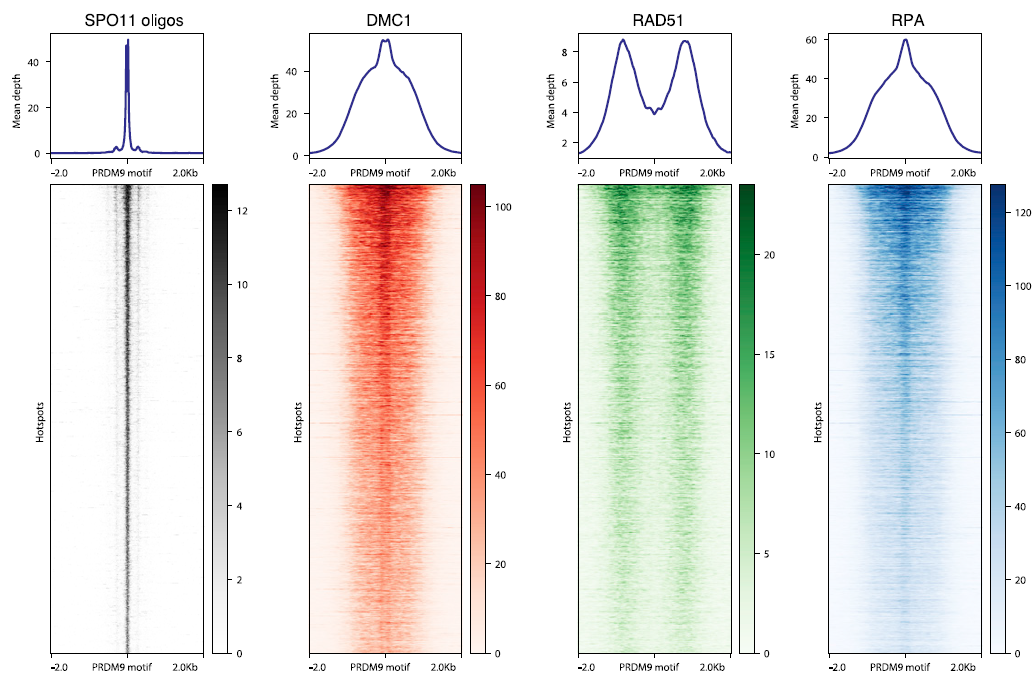
ChIP-seq数据发现损伤位点(SPO11指示)附近RPA、DMC1、RAD51有独特的分布方式:DMC1、RPA聚集到损伤处,在损伤处密度最高,而RAD51围着损伤处呈双峰分布
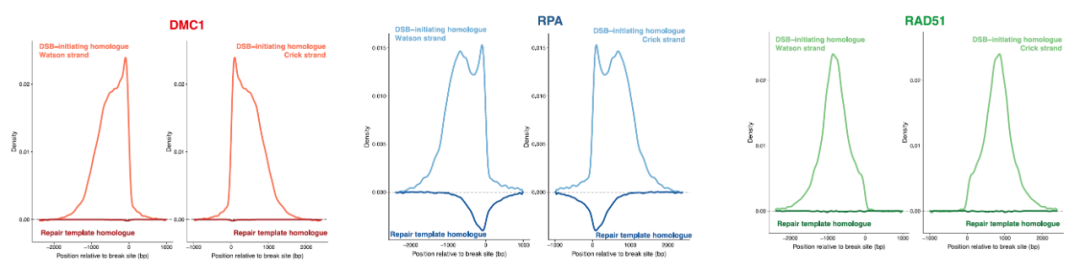
DSB后的修复依赖同源染色体作为模板,作者搭建的系统可区分两条同源染色体,即除了能看损伤链上RPA、DMC1、RAD51的分布外,还能看同源的模板链上RPA、DMC1、RAD51的分布,发现模板链上没用DMC1、RAD51分布,有RPA分布
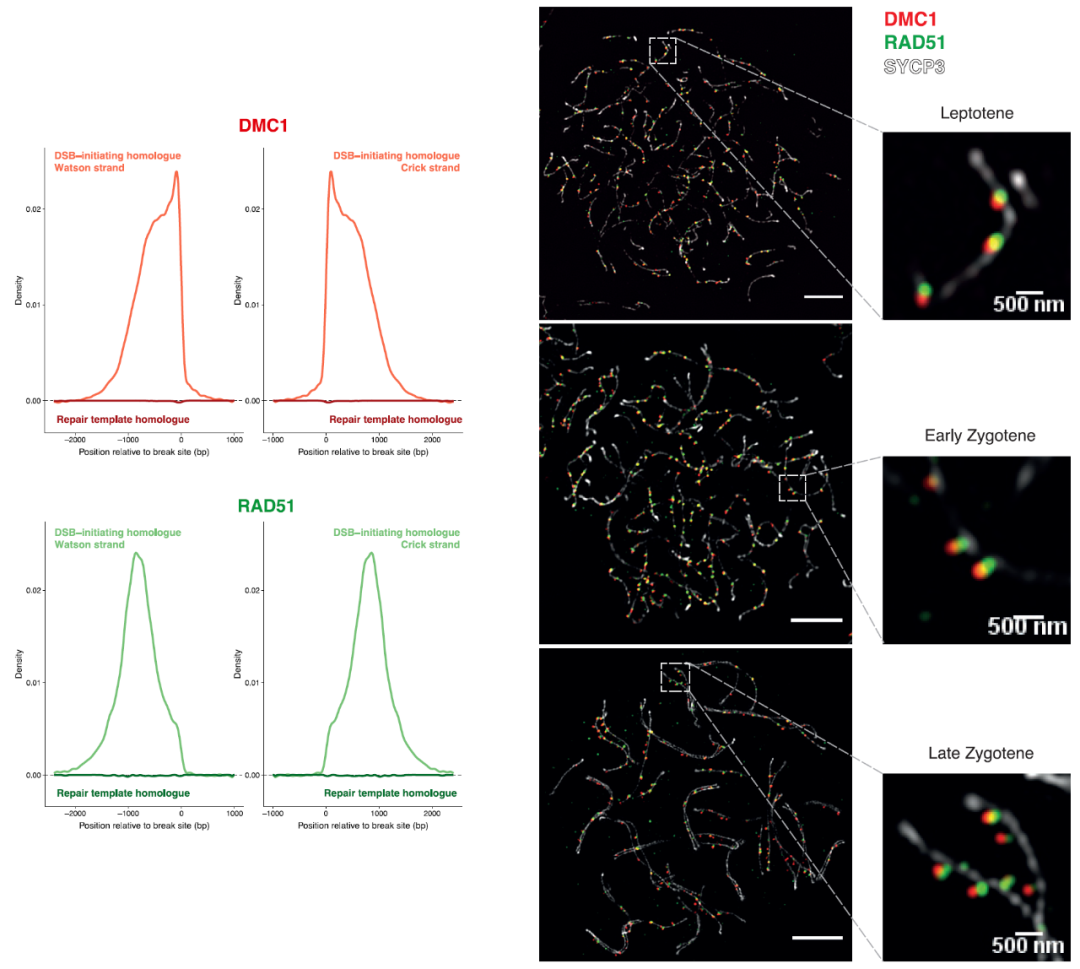
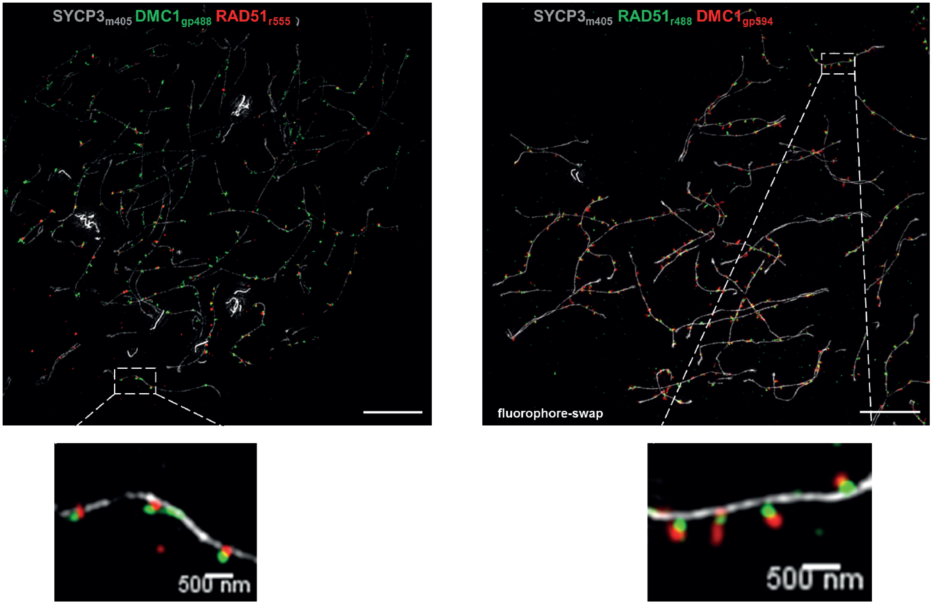
DMC1分布峰在损伤处,而RAD51分布峰距离损伤处较远,暗示两者并非完全共定位,作者实际用SIM超分辨进行了验证,发现两者的确只是部分共定位
-
瑞士乳杆菌R0052通过调节肠道微生物和代谢组减轻D-半乳糖胺诱导的肝损伤2020-01-10
-
解淀粉芽孢杆菌SC06通过调节代谢和肠道菌群,保护宿主免受高脂饮食引起的肥胖和肝损伤2019-06-27
-
NADP+是DNA损伤应答和肿瘤抑制中的内源性PARP抑制剂2019-06-05
-
酵母中反式DNA末端连接频率不受双链断裂预损伤空间邻近性的影响2019-06-05
-
可视化DNA损伤分析新技术揭示:氧化性DNA损伤在异染色质和常染色质中的修复方式是不同的2019-06-04